
以抗绿色荧光蛋白GFP纳米抗体为模型,成功地开发了多肽条形码2.0,相较于传统的低通量技术和展示技术,该方法没有来自固定效应的不利影响,不改变纳米抗体的结合特性,是一种简单、快速、定量和可靠的方法,可以同时研究数百个游离Nbs的序列功能关系。
✎ 前 言
2019年,Yusei Matsuzaki等人发表了一篇关于“Peptide barcoding for establishment of new types of genotype–phenotype linkages”的文章,文章提出了一种新的方法:肽条形码,可用于蛋白质结合物的序列-功能关系的研究。将独特的肽条形码与纳米抗体融合后与抗原包被的磁珠反应,去除非特异性纳米抗体,将功能性纳米抗体的肽条形码用特定的蛋白酶切割下来,利用蛋白质组学对肽条形码进行鉴定,结果证明了该方法的可行性。2021年,Yusei Matsuzaki等人又发表了一篇题目为“Peptide barcoding for one-pot evaluation of sequence–function relationships of nanobodies”的文章,文章改善了实验方法,利用SRMAtlas数据库,解决了之前如何以可扩展的方式设计具有高电离和片段效率的肽条形码的问题,进一步证明了肽条形码的可行性,相较于现有的噬菌体展示和酵母展示等展示技术,该方法是一种高通量方法,能同时准确研究数百个蛋白质结合物的序列-功能关系。
实验方法
首先以抗GFP纳米抗体为模型应用肽条形码,将诱变的纳米抗体库与独特的肽条形码结合起来,形成不同比例的纳米抗体-抗原复合物,通过SEC对其进行精细分馏,并通过蛋白质组学对肽条形码进行高特异性量化(图1)。
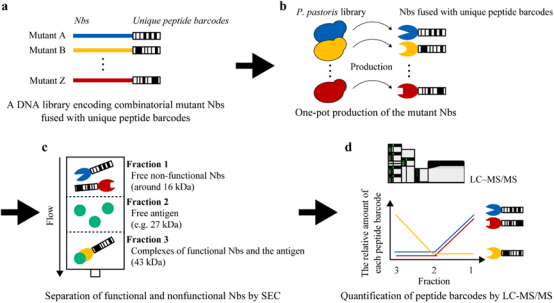
图1 肽条形码一步法评价游离纳米抗体(Nbs)的序列-功能关系
实验设计和分析
肽条形码的选择
从酵母SRMAtlas中选择候选的肽条形码,从838个候选肽中选择了107个肽,这些肽足以覆盖所有抗绿色荧光蛋白(GFP)突变纳米抗体的单个氨基酸替换。这107个多肽条码具有不同的生理化学特性,按亲水性和pI分为九类,从每一类中选择1个有代表性的多肽(图2)

图2 肽条形码的选择
将抗GFP纳米抗体与这九个有代表性的肽条形码融合,通过SPR验证融合的肽条码对抗体结合的影响。结果表明,大多数具有不同理化性质的肽条形码并不影响抗GFP纳米抗体的功能。
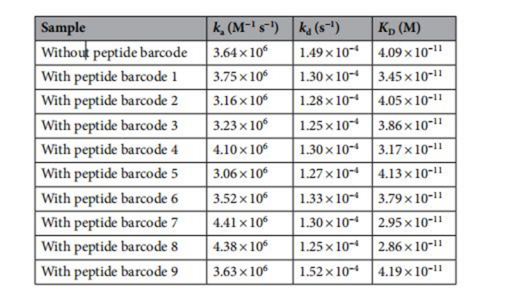
表1 抗绿色荧光蛋白野生型纳米抗体与图2c所示不同理化性质的肽条形码融合的结合动力学
生产抗GFP的丙氨酸扫描突变纳米抗体
对抗GFP的纳米抗体进行丙氨酸扫描,抗GFP纳米抗体的每个非丙氨酸突变成丙氨酸。构建一个由107个质粒组成的文库,编码抗GFP 野生型纳米抗体和抗GFP的丙氨酸扫描突变纳米抗体,并在其C端融合了独特的肽条形码,并在酵母中进行表达纯化。
利用多肽条形码技术一步法对游离的的抗GFP突变纳米抗体的序列-功能关系进行评价 采用SEC对功能性和非功能性的抗GFP突变纳米抗体进行一次性分离。收集实验中得到的这些馏分(F1-F14),并通过十二烷基硫酸钠聚丙烯酰胺凝胶电泳(SDS-PAGE)和银染色进行分析。 大多数肽条形码在F5馏分中富集,这一结果与SEC和SDS-PAGE分析一致。 为了鉴定无功能的突变体,探索含有非结合突变纳米抗体(F11-F12)的馏分中每种肽条形码的相对数量。来自五个抗GFP突变体Nbs(R35A、Y37A、W47A、G50A和E103A)的肽条形码在非结合组分中高度富集。这些结果表明,五个抗GFP突变纳米抗体对GFP的亲和力低于抗GFP WT 纳米抗体,非结合组分中每种肽条形码的相对数量的差异反映了突变纳米抗体亲和力的差异(图3)。
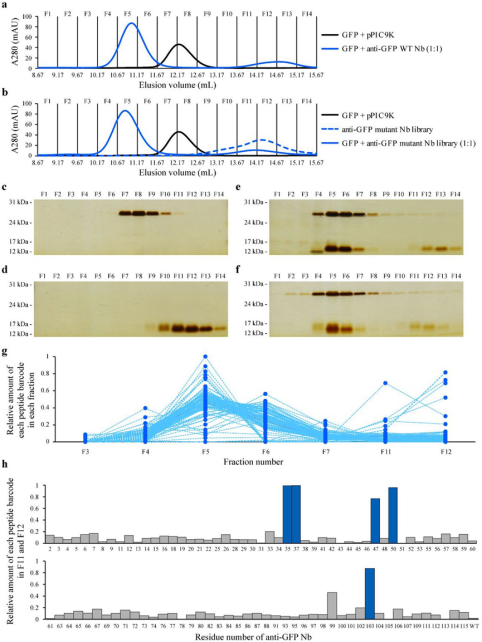
图3 对抗绿色荧光蛋白(GFP)突变体纳米抗体(Nb)库的亲和力进行一次性评估
严格富集无功能的抗GFP突变Nbs
增加GFP的量来分离低亲和力和无功能的突变纳米抗体。收集SEC分析馏分,通过SRM分析对每个馏分中的肽条形码进行定量,大多数突变纳米抗体与GFP结合并在较高分子量的馏分中被洗脱。确定F7馏分中每个肽条码的相对量,来自两个抗GFP突变体Nbs(R35A和E103A)的肽条形码在F7馏分中高度富集,而其他的肽条形码没有在F7馏分中富集。这些结果表明,R35A和E103A对GFP的亲和力非常弱或没有(图4)。

图4 通过改变绿色荧光蛋白(GFP)/和Nb的摩尔比来分离低亲和力和无功能的纳米抗体(Nbs)
通过SPR分析来验证肽条形码分析
由毕赤酵母产生五个没有肽条形码的抗GFP突变纳米抗体(R35A,Y37A,W47A,G50A和E103A),SPR分析显示,与抗GFP的WT 纳米抗体相比较,R35A、G50A和E103A不与GFP结合,Y37A和W47A的亲和力较低。这一结果与肽条形码的分析高度一致。在SEC分析中被注释为无功能性的蛋白结合物的抗GFP突变纳米抗体,在SPR分析中也不和GFP结合。在SEC分析中被注释为弱蛋白结合物的抗GFP突变纳米抗体,在SPR分析中显示出对GFP的亲和力较弱。这些结果表明,多肽条码可以多重评价游离纳米抗体的亲和力(表2)。

表2 通过肽条形码确定其亲和力下降的抗绿色荧光蛋白突变体纳米抗体,在结合动力学中表明,这些突变体没有与肽条码融合
对抗GFP Nb的序列-功能关系进行机理评估
为了验证结果,研究了抗GFP的Nb-GFP复合物的晶体结构,并计算了抗GFP的Nb-GFP复合物的结合自由能。推断出了确定的突变(R35A、Y37A、W47A、G50A和E103A)导致亲和力下降的原因,晶体结构和结合自由能分析支持了肽条形码分析的结果。
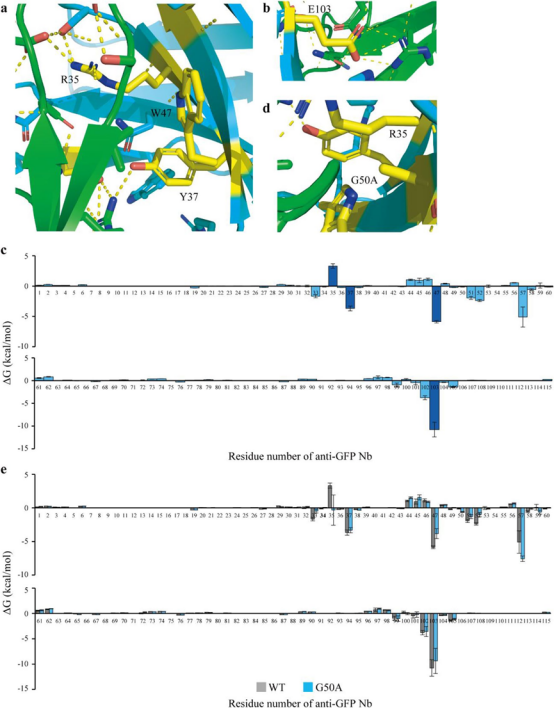
图5 重要残基对结合亲和力的影响的机理估计
✎ 结 语
以抗绿色荧光蛋白GFP纳米抗体为模型,成功地开发了多肽条形码2.0,相较于传统的低通量技术和展示技术,该方法没有来自固定效应的不利影响,不改变纳米抗体的结合特性,是一种简单、快速、定量和可靠的方法,可以同时研究数百个游离Nbs的序列功能关系。同时其具有高的检测灵敏度,可以检测出nM到sub-nM数量级的亲和力的细微差别,这往往是诊断试剂或药物所需要的,所以它也适用于纳米抗体的亲和力评估。此外,我们的方法不仅适用于蛋白质结合剂,也适用于其他类型的蛋白质,例如,它将有可能筛选出因为SEC分析中的流动性差异而无法判断的具有不同特征的蛋白质。
????文献来源:
Miyamoto Kana,Aoki Wataru,Ohtani Yuta,Miura Natsuko,Aburaya Shunsuke,Matsuzaki Yusei,Kajiwara Kaho,Kitagawa Yoshinori,Ueda Mitsuyoshi. Peptide barcoding for establishment of new types of genotype-phenotype linkages.[J]. PloS one,2019,14(4).
Matsuzaki Yusei,Aoki Wataru,Miyazaki Takumi,Aburaya Shunsuke,Ohtani Yuta,Kajiwara Kaho,Koike Naoki,Minakuchi Hiroyoshi,Miura Natsuko,Kadonosono Tetsuya,Ueda Mitsuyoshi. Peptide barcoding for one-pot evaluation of sequence–function relationships of nanobodies[J]. Scientific Reports,2021,11(1).
不感兴趣
看过了
取消

人点赞

人收藏

打赏
不感兴趣
看过了
取消






您已认证成功,可享专属会员优惠,买1年送3个月!
开通会员,资料、课程、直播、报告等海量内容免费看!

打赏金额
认可我就打赏我~
1元 5元 10元 20元 50元 其它
打赏作者
认可我就打赏我~
扫描二维码
立即打赏给Ta吧!
温馨提示:仅支持微信支付!
已收到您的咨询诉求 我们会尽快联系您
